「決定木」は機械学習のアルゴリズムの一つで、その名の通り木のような構造を持つモデルです。この記事では、決定木の視覚化について解説します。視覚化は、モデルの動作を理解し結果を解釈するための強力なツールですのでぜひご参考にしてください。
まず、決定木の視覚化を行うためには、sklearn.treeのexport_graphviz関数を使用します。この関数を使用すると、決定木の構造を.dot形式のファイルとして出力できます。このファイルを読み込むことで、決定木の視覚化が可能になります。
export_graphviz関数は、以下の主要な引数を取ります。
– `decision_tree`: 可視化したい決定木のモデルを指定します。
– `out_file`: 生成されたグラフの出力先のファイル名を指定します。
– `feature_names`: 特徴量の名前のリストを指定します。これにより、グラフ上のノードで特徴量を参照する際に、特徴量の名前が表示されます。
– `class_names`: 目的変数のクラスの名前を指定します。これにより、グラフ上のノードでクラスを参照する際に、クラスの名前が表示されます。
– `filled`: Trueに設定すると、ノードがクラスによって色分けされます。
– `rounded`: Trueに設定すると、ノードの角が丸くなります。
以下に、決定木の視覚化のための一連のコードを示します。
# 必要なライブラリのインポート
from sklearn.tree import DecisionTreeClassifier as DT
from sklearn.tree import export_graphviz
from matplotlib import pyplot as plt
from PIL import Image
import pydotplus
import io
# パラメータの設定
MAX_DEPTH = 2
RANDOM_STATE = 0
OUT_FILE = "tree.dot"
def train_decision_tree(train_X, train_y, max_depth, random_state):
# 決定木のモデル(tree)の構築と学習
tree = DT(max_depth=max_depth, random_state=random_state)
tree.fit(train_X, train_y)
return tree
def visualize_tree(tree, out_file, feature_names, class_names):
# 決定木グラフの出力
export_graphviz(
tree,
out_file=out_file,
feature_names=feature_names,
class_names=class_names,
filled=True,
rounded=True
)
# 決定木グラフの表示
g = pydotplus.graph_from_dot_file(path=out_file)
gg = g.create_png()
img = io.BytesIO(gg)
img2 = Image.open(img)
plt.figure(figsize=(img2.width/100, img2.height/100), dpi=100)
plt.imshow(img2)
plt.axis("off") # 軸の非表示
plt.show() # 画像の表示
# 決定木の学習
tree = train_decision_tree(train_X, train_y, MAX_DEPTH, RANDOM_STATE)
# 決定木の可視化
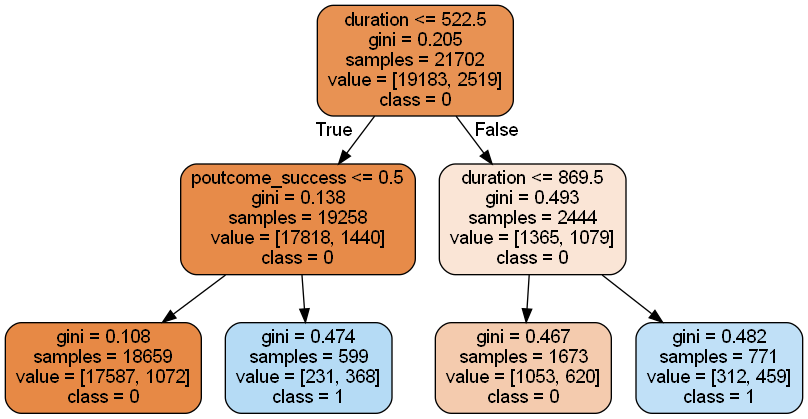
visualize_tree(tree, OUT_FILE, train_X.columns, ["0","1"])出力されるのは以下のような画像です。
import os
# PNGに変換
os.system('dot -Tpng tree.dot -o tree.png')それぞれの項目について、初めて見た時には何を意味しているのか分からないと思うので、以下説明を付しておきます。
- gini: ジニ不純度を表します。ジニ不純度は、そのノードに含まれるサンプルが純粋(すなわち、すべて同じクラスに属する)でない程度を表す指標です。ジニ不純度が0であれば、そのノードに含まれるすべてのサンプルが同じクラスに属しています。ジニ不純度が大きいほど、そのノードには複数のクラスのサンプルが混在しています。
- samples: そのノードに含まれるサンプルの総数を表します。
- value: 各クラスに属するサンプルの数を表します。例えば、二項分類の場合、value = [10, 5]は、そのノードにはクラス0に属するサンプルが10個、クラス1に属するサンプルが5個あることを意味します。
- class: そのノードに最も多くのサンプルが属するクラスを表します。つまり、そのノードが最終的にどのクラスに分類されるかを示します。
可視化してみると分岐の様子がよくわかりますよね。今回付けた図の例ではクラス1への分類がまだ奇麗にできていないように見えます(ジニ係数が高く、valueも混在している)。
このような場合は木をもう少し深くした方がよさそうですが、木を深くすると人間の目には見にくくなってきます。以下はmax_depth=6のケースです。
このような分岐を一瞬で作成してしまうのは凄いなと思いつつ、「それは結果から強引にこじつけているだけでは…」みたいな分岐もあったりして見ていると面白いです。
おわりに
あまり細かく分岐の中身まで見る機会はないかもしれませんが、特徴量の取捨やが学習の可能性などのヒントになり得ると思いますので、行き詰まった時にこのように可視化してみるのもよいかもしれません。
その時にこの記事が参考になれば幸いです。



